Difference between revisions of "Input DNA Data"
(fix <source> (syntaxhighlighting error) → <pre>) |
|||
| (70 intermediate revisions by 2 users not shown) | |||
| Line 1: | Line 1: | ||
=Input Tool= | =Input Tool= | ||
| − | [[File: | + | [[File:Main_menu_input.jpg|thumb|200px|Main menu with symbol of input new DNA data]] |
| − | This feature enables to set up references between DNA and specimen data. | + | This feature enables to set up references between DNA and specimen data. Once you have successfully logged in, click “Input Tool”. Here, specific DNA information such as: |
| − | + | * DNA extraction (e.g. extraction process, DNA quality and long-term storage) | |
| − | + | * amplified sequence fragments | |
| − | + | * respective Genbank No. and/or BOLD IDs | |
| + | can be linked to specimen data, previously integrated in a GBIF database.<br> | ||
| − | ==Specimen | + | Before entering DNA data you have to load the relevant specimen data. |
| + | |||
| + | |||
| + | <div id="wikinote">There is no possibility to save DNA data without specimen information! The DNA Module makes use of GBIF technology to reference to the underlying specimen. It is absolutely essential that voucher/specimen data are available via a GBIF compliant database! if the specimen data are not available in a GBIF compliant database, these can be set up offline with the [[Specimen Tool]].</div> | ||
| + | To guarantee both the safeguarding and long-term availability of referenced DNA samples these should be deposited in research collections. Corresponding data including voucher information have to be stored in suitable collection databases. | ||
| + | ==Specimen Details== | ||
Each DNA sample is extracted from a specimen. This specimen can be a tissue sample, a complete individual, a living plant or animal or a culture (algae, microorganisms). By defining a reference between DNA sample and the DNA voucher you should keep in mind what exactly the DNA voucher is. In terms of the DNA Bank Network the ideal DNA voucher means a complete individual, from which the tissue and DNA sample was taken from. This DNA voucher should be deposited in a natural history collection and the voucher data are available via GBIF. In many cases it is not possible to deposit such an ideal voucher, because it is for example a threatend species. Than you should reference to the most applicable DNA voucher. | Each DNA sample is extracted from a specimen. This specimen can be a tissue sample, a complete individual, a living plant or animal or a culture (algae, microorganisms). By defining a reference between DNA sample and the DNA voucher you should keep in mind what exactly the DNA voucher is. In terms of the DNA Bank Network the ideal DNA voucher means a complete individual, from which the tissue and DNA sample was taken from. This DNA voucher should be deposited in a natural history collection and the voucher data are available via GBIF. In many cases it is not possible to deposit such an ideal voucher, because it is for example a threatend species. Than you should reference to the most applicable DNA voucher. | ||
| + | [[File:InputMask.jpg|thumb|300px|Input Mask DNA Module]] | ||
===The GBIF world=== | ===The GBIF world=== | ||
| − | GBIF technologies are basis and backbone of the DNA Module and the DNA Bank Network. Many institutions are GBIF providers, more than | + | GBIF technologies are basis and backbone of the DNA Module and the DNA Bank Network. Many institutions are GBIF providers, more than 322 millions of specimen and observation records are available via the [http://data.gbif.org GBIF portal].<br> |
But how to find out if the required specimen data is available via GBIF? For that you should check the following facts: | But how to find out if the required specimen data is available via GBIF? For that you should check the following facts: | ||
* Where is the DNA voucher deposited? | * Where is the DNA voucher deposited? | ||
| − | * Is the relevant institution already a GBIF provider? Ask administrators or curators for help. | + | * Is the relevant institution already a GBIF provider? Ask administrators or curators for help or browse the GBIF portal. |
| − | + | # Enter: http://data.gbif.org/welcome.htm | |
| − | + | # Check 'Datasets' | |
| − | + | # Enter search key, e.g. 'Vienna' | |
| − | + | # At bottom of result table all relevant datasets are listed, check if the one you are looking for is listed and follow the relevant link; if you don't get useful results try another search, e.g. 'Wien' instead of 'Vienna' | |
| + | |||
| + | You should see an overview page of the relevant dataset with an occurence map etc.; at bottom you will find the Provider Url | ||
| + | |||
| + | * If so: The requirement related to specimen data is fulfilled. | ||
| + | * If not: Is the relevant institution planning or willing to become a GBIF provider? | ||
| + | ** If so: The requirement will be met if the relevant database is GBIF accessible. | ||
| + | ** If not: Relevant institution has no specimen database or no possibility of becoming a GBIF provider any time soon? Please go ahead at this point: [[Specimen Tool]] | ||
| + | |||
| + | ===Defining reference to Specimen data=== | ||
| + | To reference specimen data, the following information are required:<br> | ||
| + | * The unique specimen number (UnitID, CatalogueNumber).<br> | ||
| + | * The respective collection database, where the specimen (data) is stored. | ||
| + | |||
| + | ====Specimen number/UnitID/CatalogueNumber==== | ||
| + | [[File:GBIF record.jpg|thumb|250px|GBIF record]] | ||
| + | The UnitID (CatalogueNumber) is a unique identifier applied to a specimen in an database that is connected to GBIF. It is necessary to conduct a successful wrapper query. In an ideal world, the collection uses a definite voucher ID, which is also used for the database (e.g. the herbarium at the BGBM uses the barcodes for the herbarium vouchers as UnitIDs for the database). However, in other collections, the original voucher ID might differ from the UnitID in the database. In this case, a wrapper query for the voucher ID would fail and the user has to investigate for the accordant UnitID. | ||
| + | |||
| + | See screenshot of a GBIF record where you can find the UnitID/Catalogue number of a single record. | ||
| + | <div style="clear:both;"></div> | ||
| + | |||
| + | ====Specimen databases==== | ||
| + | The respective collection database can be selected from either the 'internal' or the 'external' dropdown menu, which include all databases currently integrated in your DNA Module. Then enter the UnitID/Catalogue Number and check 'Verify'. The data will be provided on the fly. | ||
| + | |||
| + | If the respective database is not yet integrated, it is possible to [[#Add new specimen provider|add a new specimen provider]]. | ||
| + | |||
| + | There are several cases, why a specimen record might currently not be available:<br> | ||
| + | * The respective collection has no database | ||
| + | * The respective collection has an database, but it is not accessible online via Wrapper/GBIF. | ||
| + | * The collection database is accessible online, but the wanted specimen data is not online yet. | ||
| + | * The wanted specimen is in private ownership and thus not accessible online. | ||
| + | |||
| + | In these cases, please use the [[Specimen Tool]] to add offline specimen data. These offline data can later be replaced by eventually now online available collection database. | ||
| + | |||
| + | ====Add new specimen provider==== | ||
| + | New specimen databases can be integrated with the 'New specimen provider' menu (placed at input mask. Specimen databases are generally hosted by a provider (institutes, museums, collections etc.). The provider url for each specific specimen database is generally available via GBIF (or alternatively from the respective institute). | ||
| + | [[File:New Specimen Provider01.jpg|thumb|200px|Add new specimen provider]] | ||
| + | |||
| + | This list shows three different examples of provider urls:<br> | ||
| + | Example 1: http://ww3.bgbm.org/biocase/pywrapper.cgi?dsa=Herbar<br> | ||
| + | Example 2: http://herpnet.ua.edu/DiGIRprov/DiGIR.php<br> | ||
| + | |||
| + | In following, the standard procedure to add a new specimen database via GBIF is described. Follow steps 1 to 4 for checking if an institution is a GBIF provider already. Every dataset/data provider has an overview page where you find the provider URL at the bottom. | ||
| + | [[File:New Specimen Provider02.jpg|thumb|200px|Add new specimen provider]] | ||
| + | # Copy Provider Url ('Access Point Url') and paste into the 'Wrapper Url'-Field -> Check 'Verify' | ||
| + | # If the Url does not exist yet, you can now set up a new provider/dataset. The following information have to be provided: | ||
| + | |||
| + | * '''Database scheme''' ('Schema') | ||
| + | * '''DiGIR Resource/Source''' (if using a DiGIR database) | ||
| + | * '''Display''' (choose a name for this database) | ||
| + | * '''Internal or external database''' (Internal = a database from your own institution) | ||
| + | |||
| + | The '''database scheme''' is mandatory and can be selected from a dropdown list (ABCD 1.2, ABCD 2.06, DWC (CatalogNumber), DWC (CatalogNumberText)). This information can either be found directly in the Url (in the case of Digir databases) or retrieved by accessing the Url. The latter will display an xml-scheme, where 'Supportedschemas' provides the correct scheme information.<br> | ||
| + | |||
| + | Example 1: | ||
| + | <SupportedSchemas request="true" namespace="http://www.tdwg.org/schemas/abcd/2.06" | ||
| + | response="true"> | ||
| + | Here, the ABCD 2.06 scheme is used. | ||
| + | |||
| + | '''DiGIR specials:''' | ||
| + | |||
| + | 1. If using Digir databases, the ''''Resource' and 'Source'''' information are mandatory. These can also be retrieved by accessing the url. In the header, you will find a line referring to 'source' and 'resource'.<br> | ||
| + | |||
| + | Example 2: | ||
| + | <pre> | ||
| + | <response> | ||
| + | <header> | ||
| + | <version>$Revision: 1.21 $</version> | ||
| + | <sendTime>2012-01-17 07:46:18.00Z</sendTime> | ||
| + | <source>http://herpnet.ua.edu:80/DiGIRprov/DiGIR.php</source> | ||
| + | <type>metadata</type> | ||
| + | </header> | ||
| + | </response> | ||
| + | <...> | ||
| + | <resource><name>Herp Specimens</name> | ||
| + | <code>HerpSpecimensDwC2</code> | ||
| + | <relatedInformation/> | ||
| + | </resource> | ||
| + | </pre> | ||
| + | |||
| + | Here, 'HerpSpecimensDwC2' refers to the resource and http://herpnet.ua.edu:80/DiGIRprov/DiGIR.php refers to the 'source'. Please paste these information into the respective fields. The 'source' information is mostly similar or identical to the provider url. | ||
| + | |||
| + | 2. '''CatalogNumber/CatalogNumberText''': | ||
| + | |||
| + | Unfortunately DarwinCore/DiGIR has two alternative elements for the CatalogNumber and you will have to check which one is used prior to save new dataset. | ||
| + | |||
| + | # Go to a single record of required dataset at GBIF portal | ||
| + | # Check 'Retrieve' -> 'Retrieve original record from data publisher' | ||
| + | |||
| + | <pre> | ||
| + | <record> | ||
| + | <darwin:InstitutionCode>UAHC</darwin:InstitutionCode> | ||
| + | <darwin:CollectionCode>Main</darwin:CollectionCode> | ||
| + | <darwin:CatalogNumberText>871</darwin:CatalogNumberText> | ||
| + | <darwin:ScientificName>Acris gryllus</darwin:ScientificName> | ||
| + | <...> | ||
| + | </record> | ||
| + | </pre> | ||
| + | |||
| + | Here, the provider uses CatalogNumberText, so you should select 'DWC (digir, CatalogNumberText)'. If both CatalogNumber and CatalogNumberText is used please select 'DWC (digir, CatalogNumber)'. | ||
| + | |||
| + | ==DNA Details== | ||
| + | In this section, the DNA extraction details can be linked with the respective voucher specimen. Furthermore, associated information, like amplified fragments and Genbank Acc. No. or BOLD Process IDs can be added. | ||
| + | |||
| + | The following table provides explanations and an example to all DNA details. | ||
| + | |||
| + | {| class="wikitable" | ||
| + | |- style="background-color:#C0C0C0;font-weight:bold" valign="bottom" | ||
| + | | width="166" height="13" | DNA and Tissue Data | ||
| + | | width="289" | Explanation | ||
| + | | width="57" | Pre-defined | ||
| + | | width="135" | Mandatory? | ||
| + | | width="220" | Example | ||
| + | |||
| + | |- style="font-weight:bold" | ||
| + | | height="13" valign="bottom" | General Details: | ||
| + | | valign="bottom" | | ||
| + | | valign="bottom" | | ||
| + | | valign="bottom" | | ||
| + | | align="right" valign="bottom" | | ||
| + | |||
| + | |- valign="bottom" | ||
| + | | height="13" | DNA Extraction Number | ||
| + | | A unique identifier or code for this individual DNA sample. | ||
| + | | No | ||
| + | | Yes | ||
| + | | ZFMK-DNA ColCar 0399 | ||
| + | |||
| + | |- valign="bottom" | ||
| + | | height="13" | Relation to Voucher | ||
| + | | Relation between DNA/Tissue and voucher specimen. | ||
| + | | Yes | ||
| + | | Yes | ||
| + | | DNA from specimen (voucher) | ||
| + | |||
| + | |- valign="bottom" | ||
| + | | height="13" | Tissue | ||
| + | | Type of tissue | ||
| + | | No | ||
| + | | Yes | ||
| + | | leg | ||
| + | |||
| + | |- valign="bottom" | ||
| + | | height="13" | Preservation | ||
| + | | Method of preservation | ||
| + | | Yes | ||
| + | | Yes | ||
| + | | in alcohol (ethanol, 96%) | ||
| + | |||
| + | |- valign="bottom" | ||
| + | | height="13" | DNA Type | ||
| + | | Origin of DNA | ||
| + | | Yes | ||
| + | | No | ||
| + | | gDNA | ||
| + | |||
| + | |- style="background-color:#C0C0C0" | ||
| + | |style="font-weight:bold" height="13" valign="bottom" | Extraction Details: | ||
| + | |style="font-weight:bold" valign="bottom" | | ||
| + | |style="font-weight:bold" valign="bottom" | | ||
| + | |style="font-weight:bold" valign="bottom" | | ||
| + | | align="right" valign="bottom" | | ||
| + | |||
| + | |- valign="bottom" | ||
| + | | height="13" | DNA Extraktion Date | ||
| + | | Date of DNA extraction;YYYY-MM-DD | ||
| + | | Yes | ||
| + | | Yes | ||
| + | | align="right" | | ||
| + | |||
| + | |- valign="bottom" | ||
| + | | height="13" | DNA Extraktion Method: | ||
| + | | DNA isolation kit (company/product name) or extraction protocol. | ||
| + | | No | ||
| + | | Yes; if unknown = "Unknown" | ||
| + | | Unknown | ||
| + | |||
| + | |- valign="bottom" | ||
| + | | height="13" | DNA Extraktion Staff | ||
| + | | Person who extraced DNA | ||
| + | | No | ||
| + | | Yes; if unknown = "Unknown" | ||
| + | | C.Blume/C.Etzbauer | ||
| + | |||
| + | |- style="background-color:#C0C0C0;font-weight:bold" | ||
| + | | height="13" | Quality Details: | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | | align="right" | | ||
| + | |||
| + | |- valign="bottom" | ||
| + | | height="13" | DNA Purification Method | ||
| + | | DNA purification kit (company/product name) or protocol. | ||
| + | | | ||
| + | | Yes; if unknown = "Unknown" | ||
| + | | QIAquick PCR Purification Kit Qiagen | ||
| + | |||
| + | |- valign="bottom" | ||
| + | | height="16" | Ratio of Absorbance | ||
| + | | Assessment of DNA optical density | ||
| + | | No | ||
| + | | No | ||
| + | | 1,99 OD260nm/OD280nm | ||
| + | |||
| + | |- valign="bottom" | ||
| + | | height="13" | Concentration in ng/µl | ||
| + | | Concentration of DNA | ||
| + | | No | ||
| + | | No | ||
| + | | 26,64ng/µl | ||
| + | |||
| + | |- valign="bottom" | ||
| + | | height="13" | DNA Quality | ||
| + | | Rating of DNA quality | ||
| + | | Yes | ||
| + | | No | ||
| + | | high | ||
| + | |||
| + | |- valign="bottom" | ||
| + | | height="13" | Quality Check Date | ||
| + | | Date of DNA quality check;YYYY-MM-DD | ||
| + | | Yes | ||
| + | | No | ||
| + | | - | ||
| + | |||
| + | |- style="background-color:#C0C0C0;font-weight:bold" | ||
| + | | height="13" valign="bottom" | GenBank and BOLD Entries: | ||
| + | | valign="bottom" | | ||
| + | | valign="bottom" | | ||
| + | | valign="bottom" | | ||
| + | | align="right" valign="bottom" | | ||
| + | |||
| + | |- valign="bottom" | ||
| + | | height="13" | Genetic Locus | ||
| + | | Amplified genetic locus (gene) | ||
| + | | Yes | ||
| + | | No | ||
| + | | COI | ||
| + | |||
| + | |- valign="bottom" | ||
| + | | height="13" | GenBank Acc.No / Bold Process ID | ||
| + | | Entries in Genbank or BOLD | ||
| + | | No | ||
| + | | No | ||
| + | | - | ||
| + | |||
| + | |- valign="bottom" | ||
| + | | height="13" | Link | ||
| + | | Direct links to Genbank or BOLD entries | ||
| + | | No | ||
| + | | No | ||
| + | | - | ||
| + | |||
| + | |- style="background-color:#C0C0C0;font-weight:bold" | ||
| + | | height="13" valign="bottom" | Notes: | ||
| + | | valign="bottom" | | ||
| + | | valign="bottom" | | ||
| + | | valign="bottom" | | ||
| + | | align="right" valign="bottom" | | ||
| + | |||
| + | |- valign="bottom" | ||
| + | | height="13" | DNA Sample Provided by | ||
| + | | Person who provided the DNA | ||
| + | | Yes | ||
| + | | Yes; if unknown = "Unknown" | ||
| + | | Zoological Research Museum Alexander Koenig | ||
| + | |||
| + | |- valign="bottom" | ||
| + | | height="13" | Blocked Until | ||
| + | | Sample data will be visible via web portal but can not be ordered until the given date; YYYY-MM-DD | ||
| + | | - | ||
| + | | No | ||
| + | | - | ||
| + | |||
| + | |- valign="bottom" | ||
| + | | height="13" | Remarks for Customers | ||
| + | | - | ||
| + | | - | ||
| + | | No | ||
| + | | - | ||
| + | |||
| + | |- valign="bottom" | ||
| + | | height="13" | Internal Remarks <sup>1</sup>) | ||
| + | | - | ||
| + | | - | ||
| + | | No | ||
| + | | - | ||
| + | |||
| + | |- style="background-color:#C0C0C0;font-weight:bold" | ||
| + | | height="13" valign="bottom" | Stock/Aliquots: <sup>1</sup>) | ||
| + | | valign="bottom" | | ||
| + | | valign="bottom" | | ||
| + | | valign="bottom" | | ||
| + | | align="right" valign="bottom" | | ||
| + | |||
| + | |- valign="bottom" | ||
| + | | height="13" | Fridge/Rack/Box | ||
| + | | See comments | ||
| + | | - | ||
| + | | No | ||
| + | | - | ||
| + | |||
| + | |- valign="bottom" | ||
| + | | height="13" | Barcode | ||
| + | | See comments | ||
| + | | - | ||
| + | | No | ||
| + | | - | ||
| + | |||
| + | |- valign="bottom" | ||
| + | | height="13" | Position | ||
| + | | See comments | ||
| + | | - | ||
| + | | No | ||
| + | | - | ||
| + | |||
| + | |- valign="bottom" | ||
| + | | height="13" | Source volume (µl) | ||
| + | | Original volume of aliquot/stock | ||
| + | | - | ||
| + | | No | ||
| + | | - | ||
| + | |||
| + | |- valign="bottom" | ||
| + | | height="13" | Remaining volume (µl) | ||
| + | | Volume of aliquot/stock left | ||
| + | | - | ||
| + | | No | ||
| + | | - | ||
| + | |- valign="bottom" | ||
| + | | height="13" | Price per Aliqout | ||
| + | | Defined via [[Configuration_Tool#General_Settings | Configuration Tool/General Settings]]; individual prices are possible | ||
| + | | - | ||
| + | | No | ||
| + | | - | ||
| − | + | |} | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | + | <sup>1</sup>) Not shown in the DNA Bank Network webportal. | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | === | + | ====Comments on the DNA details==== |
| − | |||
| − | + | The DNA numbers are sorted in ascending order. 'Last DNA No.' displays the highest assigned DNA number, which generally (but not always) refers to the last entered number. | |
| − | |||
| − | |||
| − | + | '''DNA Extraction Number'''<br> | |
| − | + | With the [[Configuration_Tool#General_Settings | Configuration Tool/General Settings]], institutional codes (Prefix) might can be defined for all provided data sets. | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | + | '''Relation to voucher'''<br> | |
| − | + | 'No voucher available (voucher->observation)' should be selected, if only parts of an organism (blood, feathers or leaves) have been collected. | |
| − | |||
| − | < | ||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | + | '''Tissue'''<br> | |
| − | + | 'Tissue material gone': Please select this box, if the tissue used for DNA extraction has been used up. | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | + | '''Extraction date'''<br> | |
| − | + | 'Extraction Date not available': Please select this box, if the extraction date cannot be determined any more. | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | + | '''Ratio of Absorbance'''<br> | |
| + | The ratio OD<sub>260nm/OD280nm</sub> provides an estimation of the purity of the sample. The measured value should range from 1.8 to 2.1. The ratio OD<sub>260nm/OD230nm</sub> provides an estimation of the purity against polysaccharides and polyphenol (important for some plants). The measured value should range above 2.0. | ||
| − | + | '''Genbank and BOLD entries'''<br> | |
| − | + | If available, respective Genbank No. and BOLD Process IDs can be provided here. Beside these accession numbers, the 'Link' field can be used to provide a direct link to the Genbank or BOLD entries. If you want to enter more than one entry, check the 'add Genbank Entries' box. Please don't forget to enter the '''Genetic Locus'''! | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | + | '''Blocking specimen data'''<br> | |
| + | The DNA Bank offers the possibility to block the DNA details for a limited period of time or in general. For this purpose, you can enter a date in the 'block until' field. The DNA data will be visible via web portal but cannot be ordered until the given date. Alternatively, if the DNA data should GENERALLY not be searchable or available via the DNA Bank Network's webportal, please check the box 'Block in General'. | ||
| − | + | '''Stock/Aliquots'''<br> | |
| − | + | Here specific information on the storage of stocks and aliquots of the extracted DNA can be provided. 'Fridge/Rack/Box' and 'Position in fridge' allow a detailed information on the storage placement. If a 'Barcode' is used, this can be entered in the respective field. The 'Price' for an aliquot depends on the institute, which stores the specimen/DNA and/or provides the respective database. Please refer to DNA Bank administrator of the respective institute. | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | = | + | =='Save' and 'Save + Carry Forward'== |
| + | After succesfully filling out all fields for which you can provide information, please select 'save' to add the DNA details to the respective specimen voucher, or click on 'save and carry forward' if you wish to add DNA details to more than one specimen voucher, so that you do not have to fill out all fields again. | ||
| + | If you chose 'Save + Carry Forward' a new consecutively 'Extraction No.' will automatically be generated and all fields previously filled will contain the same information as the voucher you entered before. | ||
| − | + | If you just click on 'save new specimen', the DNA details will be saved to the database and you can start with a new, empty input-sheet. | |
[[Category:DNA_Module]] | [[Category:DNA_Module]] | ||
Latest revision as of 12:39, 9 January 2023
Contents
Input Tool
This feature enables to set up references between DNA and specimen data. Once you have successfully logged in, click “Input Tool”. Here, specific DNA information such as:
- DNA extraction (e.g. extraction process, DNA quality and long-term storage)
- amplified sequence fragments
- respective Genbank No. and/or BOLD IDs
can be linked to specimen data, previously integrated in a GBIF database.
Before entering DNA data you have to load the relevant specimen data.
To guarantee both the safeguarding and long-term availability of referenced DNA samples these should be deposited in research collections. Corresponding data including voucher information have to be stored in suitable collection databases.
Specimen Details
Each DNA sample is extracted from a specimen. This specimen can be a tissue sample, a complete individual, a living plant or animal or a culture (algae, microorganisms). By defining a reference between DNA sample and the DNA voucher you should keep in mind what exactly the DNA voucher is. In terms of the DNA Bank Network the ideal DNA voucher means a complete individual, from which the tissue and DNA sample was taken from. This DNA voucher should be deposited in a natural history collection and the voucher data are available via GBIF. In many cases it is not possible to deposit such an ideal voucher, because it is for example a threatend species. Than you should reference to the most applicable DNA voucher.
The GBIF world
GBIF technologies are basis and backbone of the DNA Module and the DNA Bank Network. Many institutions are GBIF providers, more than 322 millions of specimen and observation records are available via the GBIF portal.
But how to find out if the required specimen data is available via GBIF? For that you should check the following facts:
- Where is the DNA voucher deposited?
- Is the relevant institution already a GBIF provider? Ask administrators or curators for help or browse the GBIF portal.
- Enter: http://data.gbif.org/welcome.htm
- Check 'Datasets'
- Enter search key, e.g. 'Vienna'
- At bottom of result table all relevant datasets are listed, check if the one you are looking for is listed and follow the relevant link; if you don't get useful results try another search, e.g. 'Wien' instead of 'Vienna'
You should see an overview page of the relevant dataset with an occurence map etc.; at bottom you will find the Provider Url
- If so: The requirement related to specimen data is fulfilled.
- If not: Is the relevant institution planning or willing to become a GBIF provider?
- If so: The requirement will be met if the relevant database is GBIF accessible.
- If not: Relevant institution has no specimen database or no possibility of becoming a GBIF provider any time soon? Please go ahead at this point: Specimen Tool
Defining reference to Specimen data
To reference specimen data, the following information are required:
- The unique specimen number (UnitID, CatalogueNumber).
- The respective collection database, where the specimen (data) is stored.
Specimen number/UnitID/CatalogueNumber
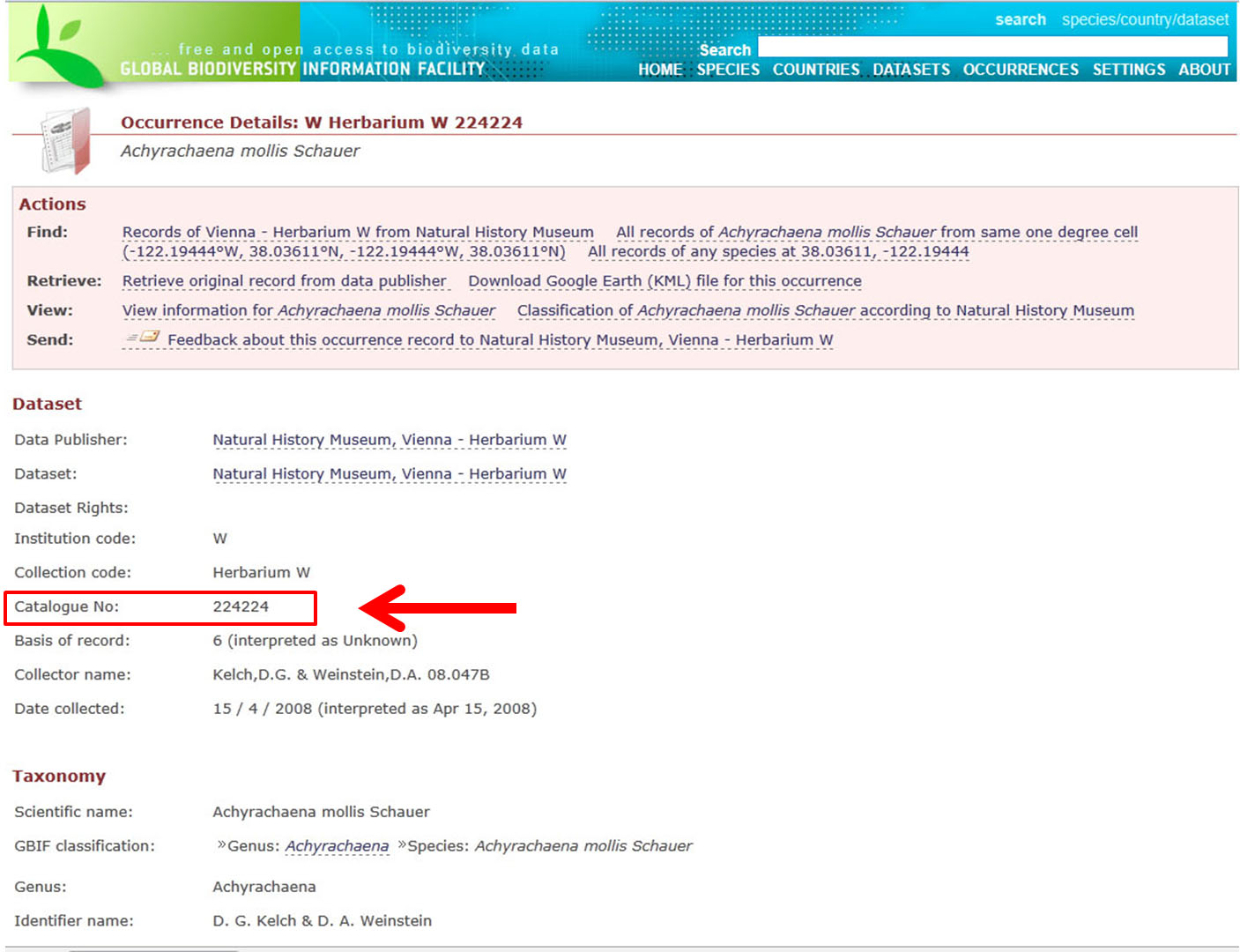
The UnitID (CatalogueNumber) is a unique identifier applied to a specimen in an database that is connected to GBIF. It is necessary to conduct a successful wrapper query. In an ideal world, the collection uses a definite voucher ID, which is also used for the database (e.g. the herbarium at the BGBM uses the barcodes for the herbarium vouchers as UnitIDs for the database). However, in other collections, the original voucher ID might differ from the UnitID in the database. In this case, a wrapper query for the voucher ID would fail and the user has to investigate for the accordant UnitID.
See screenshot of a GBIF record where you can find the UnitID/Catalogue number of a single record.
Specimen databases
The respective collection database can be selected from either the 'internal' or the 'external' dropdown menu, which include all databases currently integrated in your DNA Module. Then enter the UnitID/Catalogue Number and check 'Verify'. The data will be provided on the fly.
If the respective database is not yet integrated, it is possible to add a new specimen provider.
There are several cases, why a specimen record might currently not be available:
- The respective collection has no database
- The respective collection has an database, but it is not accessible online via Wrapper/GBIF.
- The collection database is accessible online, but the wanted specimen data is not online yet.
- The wanted specimen is in private ownership and thus not accessible online.
In these cases, please use the Specimen Tool to add offline specimen data. These offline data can later be replaced by eventually now online available collection database.
Add new specimen provider
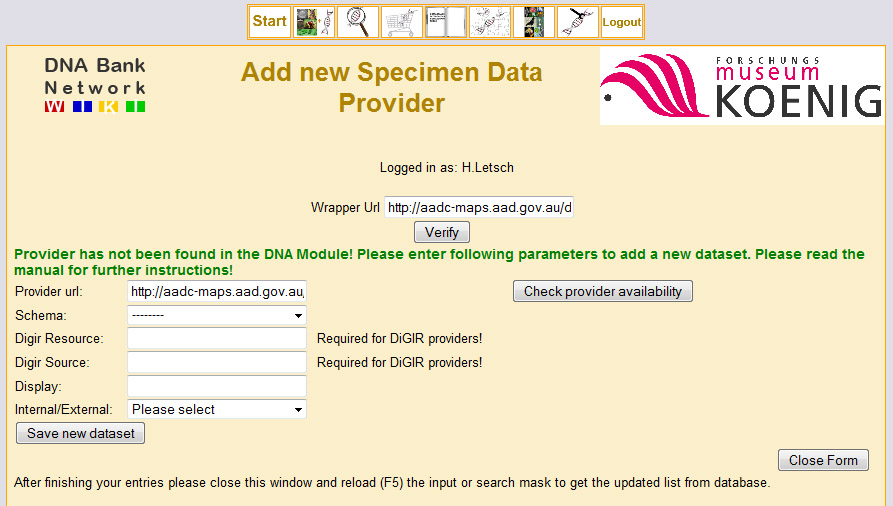
New specimen databases can be integrated with the 'New specimen provider' menu (placed at input mask. Specimen databases are generally hosted by a provider (institutes, museums, collections etc.). The provider url for each specific specimen database is generally available via GBIF (or alternatively from the respective institute).
This list shows three different examples of provider urls:
Example 1: http://ww3.bgbm.org/biocase/pywrapper.cgi?dsa=Herbar
Example 2: http://herpnet.ua.edu/DiGIRprov/DiGIR.php
In following, the standard procedure to add a new specimen database via GBIF is described. Follow steps 1 to 4 for checking if an institution is a GBIF provider already. Every dataset/data provider has an overview page where you find the provider URL at the bottom.
- Copy Provider Url ('Access Point Url') and paste into the 'Wrapper Url'-Field -> Check 'Verify'
- If the Url does not exist yet, you can now set up a new provider/dataset. The following information have to be provided:
- Database scheme ('Schema')
- DiGIR Resource/Source (if using a DiGIR database)
- Display (choose a name for this database)
- Internal or external database (Internal = a database from your own institution)
The database scheme is mandatory and can be selected from a dropdown list (ABCD 1.2, ABCD 2.06, DWC (CatalogNumber), DWC (CatalogNumberText)). This information can either be found directly in the Url (in the case of Digir databases) or retrieved by accessing the Url. The latter will display an xml-scheme, where 'Supportedschemas' provides the correct scheme information.
Example 1:
<SupportedSchemas request="true" namespace="http://www.tdwg.org/schemas/abcd/2.06" response="true">
Here, the ABCD 2.06 scheme is used.
DiGIR specials:
1. If using Digir databases, the 'Resource' and 'Source' information are mandatory. These can also be retrieved by accessing the url. In the header, you will find a line referring to 'source' and 'resource'.
Example 2:
<response> <header> <version>$Revision: 1.21 $</version> <sendTime>2012-01-17 07:46:18.00Z</sendTime> <source>http://herpnet.ua.edu:80/DiGIRprov/DiGIR.php</source> <type>metadata</type> </header> </response> <...> <resource><name>Herp Specimens</name> <code>HerpSpecimensDwC2</code> <relatedInformation/> </resource>
Here, 'HerpSpecimensDwC2' refers to the resource and http://herpnet.ua.edu:80/DiGIRprov/DiGIR.php refers to the 'source'. Please paste these information into the respective fields. The 'source' information is mostly similar or identical to the provider url.
2. CatalogNumber/CatalogNumberText:
Unfortunately DarwinCore/DiGIR has two alternative elements for the CatalogNumber and you will have to check which one is used prior to save new dataset.
- Go to a single record of required dataset at GBIF portal
- Check 'Retrieve' -> 'Retrieve original record from data publisher'
<record>
<darwin:InstitutionCode>UAHC</darwin:InstitutionCode>
<darwin:CollectionCode>Main</darwin:CollectionCode>
<darwin:CatalogNumberText>871</darwin:CatalogNumberText>
<darwin:ScientificName>Acris gryllus</darwin:ScientificName>
<...>
</record>
Here, the provider uses CatalogNumberText, so you should select 'DWC (digir, CatalogNumberText)'. If both CatalogNumber and CatalogNumberText is used please select 'DWC (digir, CatalogNumber)'.
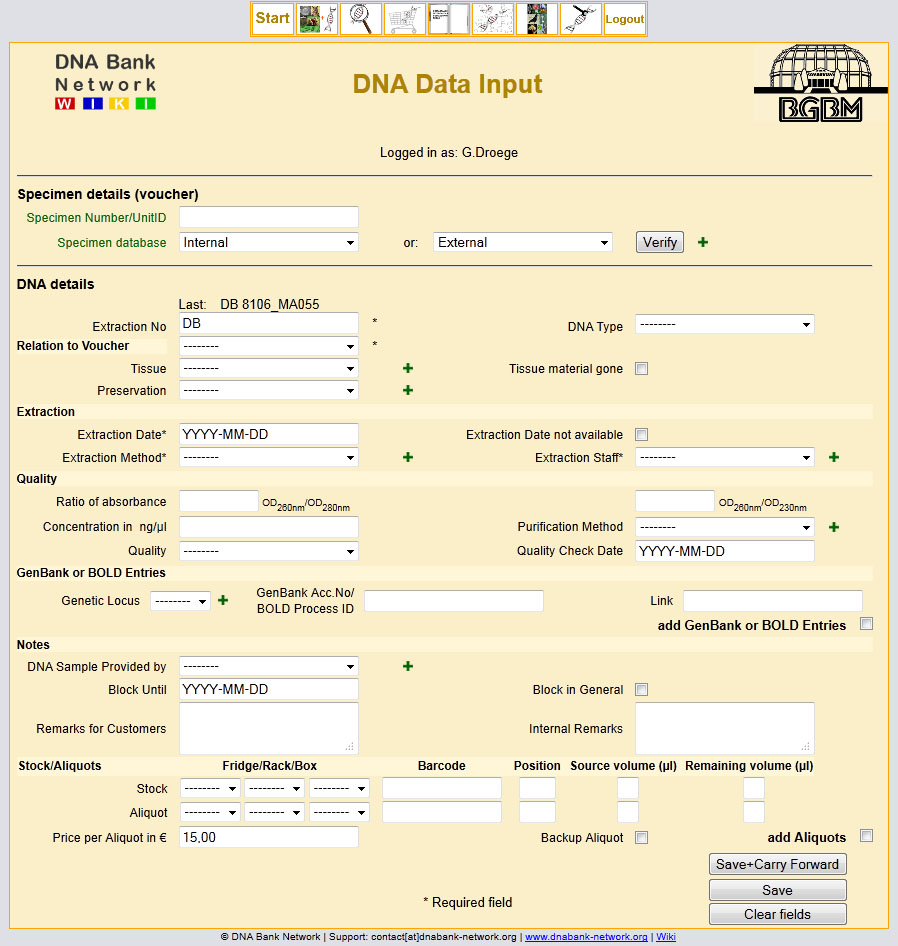
DNA Details
In this section, the DNA extraction details can be linked with the respective voucher specimen. Furthermore, associated information, like amplified fragments and Genbank Acc. No. or BOLD Process IDs can be added.
The following table provides explanations and an example to all DNA details.
| DNA and Tissue Data | Explanation | Pre-defined | Mandatory? | Example |
| General Details: | ||||
| DNA Extraction Number | A unique identifier or code for this individual DNA sample. | No | Yes | ZFMK-DNA ColCar 0399 |
| Relation to Voucher | Relation between DNA/Tissue and voucher specimen. | Yes | Yes | DNA from specimen (voucher) |
| Tissue | Type of tissue | No | Yes | leg |
| Preservation | Method of preservation | Yes | Yes | in alcohol (ethanol, 96%) |
| DNA Type | Origin of DNA | Yes | No | gDNA |
| Extraction Details: | ||||
| DNA Extraktion Date | Date of DNA extraction;YYYY-MM-DD | Yes | Yes | |
| DNA Extraktion Method: | DNA isolation kit (company/product name) or extraction protocol. | No | Yes; if unknown = "Unknown" | Unknown |
| DNA Extraktion Staff | Person who extraced DNA | No | Yes; if unknown = "Unknown" | C.Blume/C.Etzbauer |
| Quality Details: | ||||
| DNA Purification Method | DNA purification kit (company/product name) or protocol. | Yes; if unknown = "Unknown" | QIAquick PCR Purification Kit Qiagen | |
| Ratio of Absorbance | Assessment of DNA optical density | No | No | 1,99 OD260nm/OD280nm |
| Concentration in ng/µl | Concentration of DNA | No | No | 26,64ng/µl |
| DNA Quality | Rating of DNA quality | Yes | No | high |
| Quality Check Date | Date of DNA quality check;YYYY-MM-DD | Yes | No | - |
| GenBank and BOLD Entries: | ||||
| Genetic Locus | Amplified genetic locus (gene) | Yes | No | COI |
| GenBank Acc.No / Bold Process ID | Entries in Genbank or BOLD | No | No | - |
| Link | Direct links to Genbank or BOLD entries | No | No | - |
| Notes: | ||||
| DNA Sample Provided by | Person who provided the DNA | Yes | Yes; if unknown = "Unknown" | Zoological Research Museum Alexander Koenig |
| Blocked Until | Sample data will be visible via web portal but can not be ordered until the given date; YYYY-MM-DD | - | No | - |
| Remarks for Customers | - | - | No | - |
| Internal Remarks 1) | - | - | No | - |
| Stock/Aliquots: 1) | ||||
| Fridge/Rack/Box | See comments | - | No | - |
| Barcode | See comments | - | No | - |
| Position | See comments | - | No | - |
| Source volume (µl) | Original volume of aliquot/stock | - | No | - |
| Remaining volume (µl) | Volume of aliquot/stock left | - | No | - |
| Price per Aliqout | Defined via Configuration Tool/General Settings; individual prices are possible | - | No | - |
1) Not shown in the DNA Bank Network webportal.
Comments on the DNA details
The DNA numbers are sorted in ascending order. 'Last DNA No.' displays the highest assigned DNA number, which generally (but not always) refers to the last entered number.
DNA Extraction Number
With the Configuration Tool/General Settings, institutional codes (Prefix) might can be defined for all provided data sets.
Relation to voucher
'No voucher available (voucher->observation)' should be selected, if only parts of an organism (blood, feathers or leaves) have been collected.
Tissue
'Tissue material gone': Please select this box, if the tissue used for DNA extraction has been used up.
Extraction date
'Extraction Date not available': Please select this box, if the extraction date cannot be determined any more.
Ratio of Absorbance
The ratio OD260nm/OD280nm provides an estimation of the purity of the sample. The measured value should range from 1.8 to 2.1. The ratio OD260nm/OD230nm provides an estimation of the purity against polysaccharides and polyphenol (important for some plants). The measured value should range above 2.0.
Genbank and BOLD entries
If available, respective Genbank No. and BOLD Process IDs can be provided here. Beside these accession numbers, the 'Link' field can be used to provide a direct link to the Genbank or BOLD entries. If you want to enter more than one entry, check the 'add Genbank Entries' box. Please don't forget to enter the Genetic Locus!
Blocking specimen data
The DNA Bank offers the possibility to block the DNA details for a limited period of time or in general. For this purpose, you can enter a date in the 'block until' field. The DNA data will be visible via web portal but cannot be ordered until the given date. Alternatively, if the DNA data should GENERALLY not be searchable or available via the DNA Bank Network's webportal, please check the box 'Block in General'.
Stock/Aliquots
Here specific information on the storage of stocks and aliquots of the extracted DNA can be provided. 'Fridge/Rack/Box' and 'Position in fridge' allow a detailed information on the storage placement. If a 'Barcode' is used, this can be entered in the respective field. The 'Price' for an aliquot depends on the institute, which stores the specimen/DNA and/or provides the respective database. Please refer to DNA Bank administrator of the respective institute.
'Save' and 'Save + Carry Forward'
After succesfully filling out all fields for which you can provide information, please select 'save' to add the DNA details to the respective specimen voucher, or click on 'save and carry forward' if you wish to add DNA details to more than one specimen voucher, so that you do not have to fill out all fields again.
If you chose 'Save + Carry Forward' a new consecutively 'Extraction No.' will automatically be generated and all fields previously filled will contain the same information as the voucher you entered before.
If you just click on 'save new specimen', the DNA details will be saved to the database and you can start with a new, empty input-sheet.